2025年5月22日,北京大学现代农学院杨建国研究员与崖州湾国家实验室王忆平教授合作在Nucleic Acids Research期刊上发表题为Expanding the σ54-dependent transcription process with orthogonal designs的研究论文,提出并表征了一种改造细菌σ54因子实现正交转录系统的方法。
基于σ54因子的正交转录系统原理及优势示意图
在合成生物学的实践中,细菌宿主自身的遗传机器为外源基因线路的运行提供动力,并从底层上控制着预期生物学功能的执行。尽管合成生物学研究与应用所涉及的范围极其广泛,但导入宿主中的元件及模块的稳定运行始终是领域内的共性需求,其中可靠的转录行为起着首要且关键的作用。创制新型细菌正交转录系统将有助于丰富现有的合成生物学工具箱,为非模式细菌中提供正交化基因表达提供新策略,保障复杂基因线路的长期稳定运行。
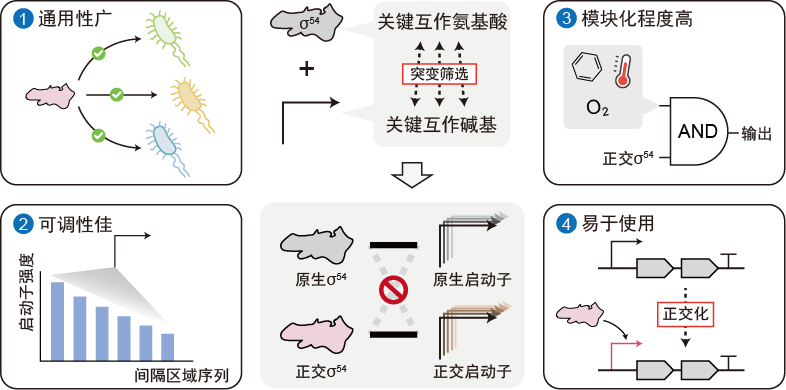
σ54因子是细菌RNA聚合酶(RNAP)中的一个可解离亚基,在RNAP全酶中负责与启动子的特异性识别与结合。通过对σ54-启动子复合体结构的分析,推测σ54的R455和R456两个氨基酸残基可通过氢键参与启动子的−24区相互作用。利用文库vs文库筛选法,得到了一个正交的σ54因子变体σ54-R456H。正交性测试实验表明,σ54-R456H仅可特异性识别与之适配的正交启动子,而失去了识别宿主自身启动子的能力。结合饱和突变、文库筛选等方法,后续发现σ54-R456H,R456Y和R456L可在细胞内同时使用,构成一组互相正交的σ因子组。考虑到σ54的R456位点在不同细菌类群中的保守性,将相同突变体导入3种非模式细菌底盘中后,均能够在其中实现正交转录,表明这套正交转录系统可在遗传背景各异的诸多细菌中发挥功能。进一步将不同类型的上游激活蛋白(bacterial Enhancer Binding Protein,bEBP)载入该系统,可实现对环境信号(如温度)或化学信号(如氧气浓度)的响应,实现依赖于特定信号的下游正交转录输出。上述结果表明,该正交转录系统本身是高度模块化的,在非模式细菌中均能发挥理想的效果;且其内部也是模块化的,bEBP可自由调整以实现更智能的自动调控。研究进一步测试了该系统用于定制正交的复杂生物学功能的潜力。选取蔗糖代谢途径、生物固氮、逻辑门等作为应用场景,使用σ54-R456H适配的正交启动子控制相应基因的转录,发现仅可在该正交σ54因子存在时,下游的基因才可开始转录,产生预期的生物学表型。这些实验表明,使用该系统可以方便地对基因线路进行正交化改造,即便对于6操纵子18基因的极复杂的生物固氮基因簇,该系统也可完全可以胜任。
综上所述,本研究通过对σ54与其启动子的识别过程进行改造,获得了三套新的σ54识别偏好性,它们与宿主自身的转录机器互相正交。基于σ54因子的正交转录系统具有通用性广、可调性佳、模块化程度高、易于使用等优点,为实现正交基因表达提供了实用的补充方案,有助于在非模式菌株中实现多基因系统的组合控制及收敛调控,也为在根系微生物中实现高效联合固氮提供了重要保障。
北京大学现代农学院杨建国研究员和崖州湾国家实验室王忆平教授为本论文共同通讯作者,北京大学前沿交叉学科研究院2020级PTN项目博士生刘逸珩为论文第一作者。北京大学现代农学院博士生蔡舒怡、张子奕、谢卓婷以及北京大学生命科学学院博士生郭琛月参与了本研究。该研究得到了国家重点研发计划项目、国家自然科学基金、农业生物育种国家科技重大专项、基因功能研究与操控全国重点实验室、崖州湾国家实验室以及北京大学现代农学院的资助。
杨建国课题组常年招收博士研究生及博士后,欢迎对生物固氮以及合成生物学感兴趣的同学加入课题组!

